- 品牌
- 有梦生物
- 服务项目
- 扫描
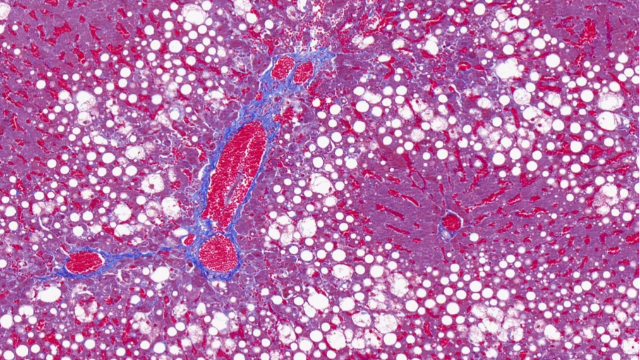
0. 植物病理学借助全景扫描技术观察病原体入侵植物的全过程,通过标记病原体与植物细胞的特异性分子,追踪病原体从附着植物表面到侵入细胞、在植物体内扩散的路径,记录植物细胞的防御反应如细胞壁加厚、植保素合成等动态变化。结合转录组学分析,揭示植物与病原体的相互作用机制,例如在研究小麦锈病时,全景扫描清晰展示了锈菌孢子的萌发、菌丝的生长及对小麦叶片细胞的破坏过程,为培育抗病品种提供了靶点,同时也为制定病害防控措施提供了科学依据。对苔藓植物群落全景扫描,探究其在岩石表面的定植与土壤形成。湖北芯片全景扫描价格实惠
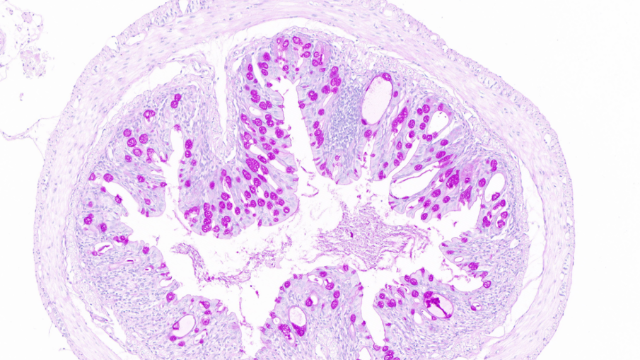
在植物化学生态学研究领域,全景扫描技术凭借成像技术与高精度化学分析的深度融合,成为解析植物次生代谢产物动态机制的关键工具。该技术不仅能精细捕捉代谢产物在植物体内的空间分布特征,还能追踪其从合成部位向体表或环境释放的全过程,为揭示植物与生物环境的化学互作提供了可视化证据。以***化感作用研究为例,通过全景扫描技术的高分辨率成像,研究者清晰观察到尼古丁在叶片表面呈现沿叶脉富集的梯度分布,并结合行为学实验证实这种分布模式与对***天蛾等害虫的驱避强度直接相关 —— 叶片边缘的高浓度尼古丁区域能***降低害虫取食频率。此类发现不仅阐明了次生代谢产物的防御策略与其空间分布的协同进化关系,更为靶向设计植物源农药提供了重要线索,例如通过调控代谢产物的合成与运输路径,增强作物的天然抗虫能力,从而减少化学农药的依赖。湖北芯片全景扫描价格实惠病毒蛋白质组学研究运用全景扫描技术结合蛋白质组学方法。
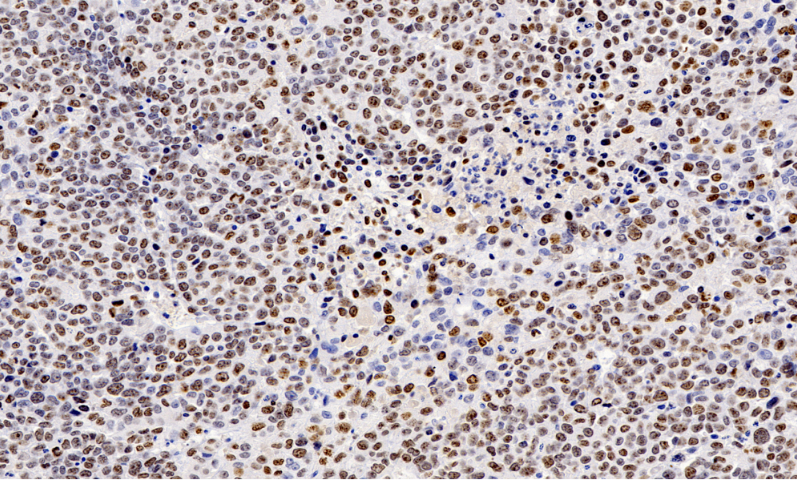
在植物发育生物学研究中,全景扫描技术实现了对植物形态建成的动态、立体化解析。通过激光共聚焦显微镜结合光学投影断层成像(OPT),研究者能够以微米级分辨率连续记录根尖分生组织细胞的不对称分裂、叶原基的极性建立以及花***的三维形态发生全过程。以模式植物拟南芥为例,全景扫描技术成功捕捉到从花序分生组织到四轮花***(萼片、花瓣、雄蕊、心皮)的渐进式发育过程,并通过荧光报告基因实时显示WUS、CLV3、AG等关键基因的表达域动态变化。该技术与单细胞转录组测序的联用,进一步构建了植物***发生的时空基因调控网络。研究发现,茎尖分生组织中细胞分裂素梯度与生长素极性运输共同决定了叶序模式(如螺旋式或对生排列)。在作物改良方面,基于全景扫描获得的水稻穗分枝三维模型,科学家精细定位了控制穗粒数的DEP1基因表达位点,为CRISPR基因编辑提供了明确靶标。此外,通过比较野生型与突变体的根系全景扫描数据,发现了PLT转录因子梯度对根冠分化的调控作用,这一发现已被应用于设计抗旱转基因作物。
在软骨组织工程研究中,全景扫描技术已成为评估工程化软骨构建质量的金标准。该技术通过多尺度成像系统实现了对软骨再生全过程的动态监控,具体包括:①微米CT(μ-CT)定量分析PCL/胶原复合支架的孔隙连通性(比较好孔径150-300μm);②双光子显微镜***追踪MSCs细胞在支架内的迁移路径与分化轨迹(SOX9、COL2A1表达);③拉曼光谱成像无标记检测GAGs和II型胶原的空间沉积规律。***研究表明,通过时间序列全景扫描发现:当支架降解速率(如PLGA)与软骨基质分泌速率达到1:1.2时,可形成比较好的力学性能(压缩模量≥0.8MPa)。这一发现直接优化了"梯度降解支架"的设计——表层快速降解诱导细胞增殖,**层缓释TGF-β3促进分化。在临床转化中,结合AI图像分析算法的全景扫描系统,可自动识别工程化软骨的纤维化区域(COLI/II比值>0.3),使产品质量控制效率提升5倍。目前,该技术已成功应用于耳廓再生和关节软骨修复,患者术后1年的T2-mapping磁共振显示,新生软骨与天然软骨的各向异性指数差异<15%。未来,整合力学-化学耦合全景扫描的新一代评估平台,将进一步推动个性化软骨组织工程产品的临床应用。
全景扫描评估人工心脏瓣膜,检测其与血液接触后的血栓形成风险。
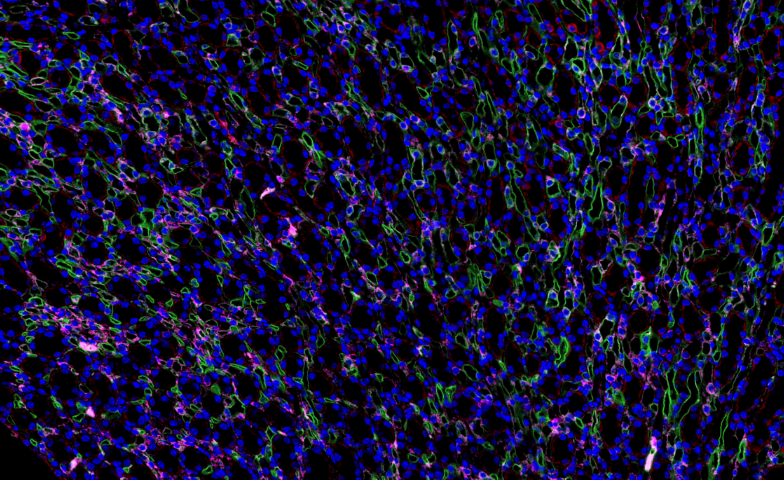
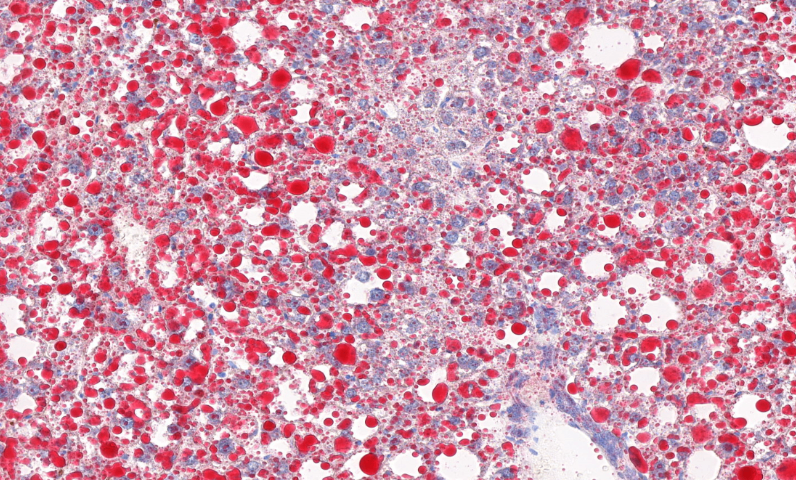
在植物逆境生理学研究中,全景扫描技术 通过多维度表型组-生理组联合分析,系统揭示了植物应对环境胁迫的适应性策略。该技术整合 高光谱成像(400-2500nm)、激光共聚焦显微术 和 X射线断层扫描,实现了从***到细胞水平的动态响应监测。以小麦抗旱研究为例,根系原位全景扫描 显示:在土壤含水量降至12%时,抗旱品种能快速启动 "深根系化" 策略(主根伸长速率提高3倍),并通过 根冠黏液层增厚(扫描电镜显示厚度增加50μm)减少水分流失。全景扫描监测叶片衰老,记录叶绿素降解与细胞结构解体的顺序。天津荧光单标全景扫描咨询报价
用全景扫描研究噬菌体疗法,观察其准确裂解致病菌的全过程。湖北芯片全景扫描价格实惠
在噬菌体研究中,全景扫描技术 通过超高时空分辨率成像系统,实现了对 噬菌体-细菌互作 全过程的动态可视化。该技术整合 冷冻电镜单颗粒分析(分辨率达2.8Å)、高速原子力显微镜(HS-AFM,毫秒级动态捕捉)和 荧光标记示踪,可解析从 初始吸附 到 裂解释放 的分子细节:侵染起始阶段冷冻电镜全景重构 显示T4噬菌体尾丝蛋白gp37通过 三聚体前列结构域(残基Asp1021-Glu1098)特异性识别大肠杆菌OmpC孔蛋白的 表面环状区(L3 loop)高速AFM动态扫描 发现噬菌体λ的J蛋白在10秒内完成 宿主Lamb受体的多点锚定(结合力≥50pN)基因组注入机制荧光量子点标记 的全景追踪显示,T7噬菌体DNA以 5kb/秒的速度 通过收缩的尾鞘注入细胞,伴随宿主 质子动力势(Δψ)的瞬时崩溃同步辐射X射线成像 捕获到噬菌体Φ29的 portal蛋白旋转(每秒120转),驱动DNA穿越细胞膜抗性突破策略超分辨显微镜(STORM)发现,CRISPR-Cas9抗性菌株的 胞内噬菌体衣壳 会*** SOS响应系统,通过RecA蛋白介导的 原噬菌体*** 逃逸切割湖北芯片全景扫描价格实惠
- 吉林TRAP染色全景扫描大概多少钱 2025-12-30
- 北京刚果红染色全景扫描单价 2025-12-30
- 黑龙江天狼猩红全景扫描大概价格 2025-12-29
- 贵州刚果红染色全景扫描大概费用 2025-12-26
- 中国香港脑组织全景扫描大概费用 2025-12-26
- 中国台湾芯片全景扫描性价比 2025-12-24
- 新疆天狼猩红全景扫描一般多少钱 2025-12-23
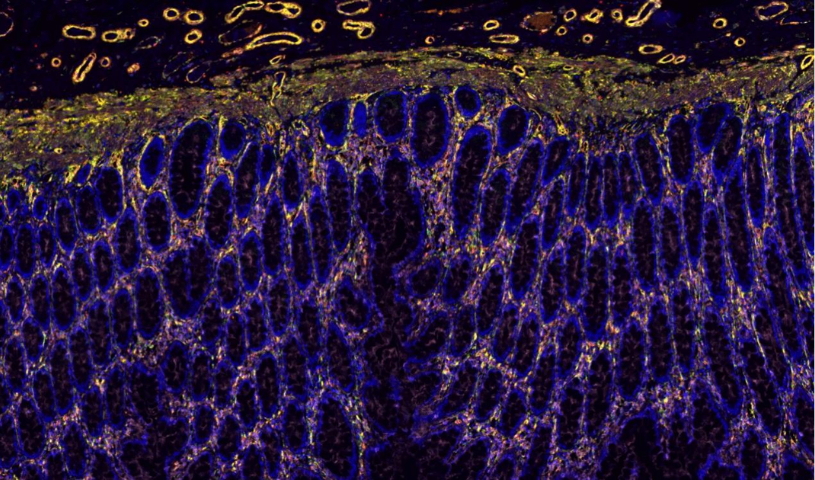
- 荧光双标全景扫描售价 2025-12-23
- 重庆荧光全景扫描一般多少钱 2025-12-22
- 浙江荧光双标全景扫描销售电话 2025-12-22
- 江西荧光多标全景扫描市场价格 2025-12-19
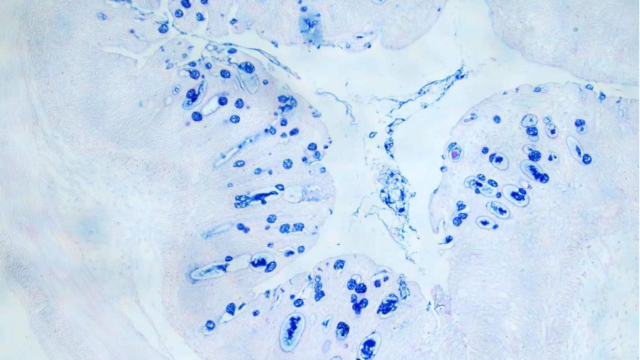
- 北京甲苯胺蓝全景扫描售价 2025-12-19